Rybonukleaza T 1
Rybonukleaza T 1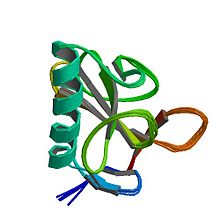 RNaza T 1 z Aspergillus oryzae ( PDB 1YGW )
RNaza T 1 z Aspergillus oryzae ( PDB 1YGW )
| Nr WE | WE |
|---|---|
| numer CAS |
| IUBMB | Wpis IUBMB |
|---|---|
| IntEnz | Widok IntEnz |
| BRENDA | Wejście BRENDA |
| KEGG | Wejście KEGG |
| MetaCyc | Szlak metaboliczny |
| PRIAM | Profil |
| PDB | Struktury |
| UDAĆ SIĘ | AmiGO / EGO |
Rybonukleazy T 1 jest endonukleaza grzybicze który katalizuje ten rozszczepienie z RNA, pozostającego w wyniku pozostałości z guanozyny , to znaczy w swoim 3 „boku . Najbardziej znana odmiana tego enzymu jest ten pochodzący z Aspergillus oryzae , z formy . Ze względu na swoją specyficzność wobec reszt guanozyny, RNaza T 1 jest często stosowana do trawienia zdenaturowanego RNA przed jego sekwencjonowaniem . Podobnie jak barnaza i rybonukleaza A , rybonukleaza T 1 była przedmiotem wielu badań nad fałdowaniem białek .
RNazy T 1 jest małe białko α + β 104 pozostałości z aminokwasów posiadające od ulotekWykonujemy p cztery wątki obejmujących długi alfa helisy (około pięć obrotów). Ma dwa mostki dwusiarczkowe między parami reszt cysteiny Cys2-Cys10 i Cys6-Cys103, z których drugi dodatkowo przyczynia się do stabilności fałdowania białek. Zakończeniu redukcji dwóch dwusiarczków ma zasadniczo wpływu rozkładania białek, chociaż na gięciu może zostać przywrócona w wysokiej soli stężeniu .
Uwagi i odniesienia
- (w) Stefania Pfeiffer, Yasmin Karimi-Nejad i Heinz Rüterjans , „ Granice określenia struktury NMR przy użyciu obliczeń zmiennej funkcji docelowej: rybonukleaza T 1 , studium przypadku ” , Journal of Molecular Biology , vol. 266 n O 2 21 lutego 1997, s. 400–423 ( PMID 9047372 , DOI 10.1006 / jmbi.1996.0784 , czytaj online )
- (w) C. Nick Pace, Udo Heinemann, Ulrich Hahn i Wolfram Saenger , „ Ribonuclease T 1 : Structure, Function, and Stability ” , Angewandte Chemie International Edition in English , vol. 30 N O 4, Kwiecień 1991, s. 343-360 ( DOI 10.1002 / anie.199103433 , czytaj online )
- (w) CN Pace, GR Grimsley, JA Thomson i BJ Barnett , „ Stabilność konformacyjna i aktywność rybonukleazy T 1 z nienaruszonymi zerowymi, jednym i dwoma wiązaniami disiarczkowymi ” , Journal of Biological Chemistry , tom. 263 n O 24, 25 sierpnia 1988, s. 11820-11825 ( PMID 2457027 , czytaj online )
- (en) Motohisa Oobatake, Sho Takahashi i Tatsuo Ooi , „ Conformational Stability of Ribonuclease T 1 . II. Renaturacja indukowana solą1 ” , Journal of Biochemistry , tom. 86, n o 1, Lipiec 1979, s. 65-70 ( PMID 113396 , czytaj online )