Phage phiX174
Phage phiX174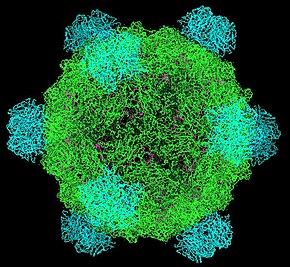 Struktura kapsydu faga geX174
Struktura kapsydu faga geX174
| Rodzaj | Wirus |
|---|---|
| Grupa | Grupa II |
| Rodzina | Microviridae |
| Uprzejmy | Sinsheimervirus |
- autor niekompletny - , data do określenia
PhiX174 fagi (lub ΦX174 ) jest wirus bakteriofaga rodziny Microviridae . Jest to bardzo prosty wirus, którego genom składa się z kolistej jednoniciowej cząsteczki DNA o długości 5386 nukleotydów i zawierającej jedenaście genów . Wirus ten został wyizolowany w 1935 roku przez Nicolasa Boulgakova (brata pisarza Mikhaïla Boulgakova ) w laboratorium Félix d'Hérelle w Institut Pasteur , z próbek pobranych w kanałach Paryża . Jego charakterystyka i badanie mechanizmu replikacji były prowadzone od lat pięćdziesiątych XX wieku, w szczególności przez Roberta Sinsheimera z Caltech w Kalifornii i Waltera Fiersa (in) z Uniwersytetu w Gandawie w Belgii .
Jednoniciowy charakter genomu ΦX174 umożliwił Frederickowi Sangerowi i jego współpracownikom przeprowadzenie sekwencjonowania już w 1977 r. , Dzięki czemu wirus ten stał się pierwszym organizmem, którego sekwencja genomowa była znana. Za tę pracę Sanger otrzymał drugą Nagrodę Nobla w dziedzinie chemii w 1980 roku.
W 2003 r. Genom ϕX174 został sztucznie zrekonstruowany przez zespół Craiga Ventera poprzez złożenie oligonukleotydów uzyskanych w wyniku syntezy chemicznej. Stwierdzono, że tak otrzymany syntetyczny wirus jest funkcjonalny, co stanowi pierwszy krok w kierunku nowej inżynierii organizmów żywych.
Białka jest wytwarzana przez phiX174 E pozwala rozbić błonę komórkową z Escherichia coli przez zjawisko zwane liza powoduje zniszczenie komórek .
Uwagi i odniesienia
- | langue = pl | "Synteza, charakterystyka, właściwości biobójcze i wirusobójcze metali ...", Johanna A.Haggstrom https://books.google.fr/booksid=v8_WMHzxZekC&pg=PA212&lpg=PA212&dq=phage+phix174+date+ de + d% C3% A9couverte & source = bl & ots = lwb8q6cu87 & sig = 2OEPYvcIvIRrOnCmeJc1-mpi5P4 & hl = fr & sa = X & ei = UWV3VOX1KYrcatfogcAG & ved = 0CDsQ6AEwBw
- (w) Sertic V., N. Bułhakow, „ Klasyfikacja i identyfikacja faga-typhi. » , Compt. robi. soc. biol. , vol. 119,1935, s. 1270-1272
- (w) Sinsheimer RL, „ Niektóre właściwości bakteriofaga φX174. " , Federation Proc. , vol. 16,1957, s. 250
- (w) Fiers W., Sinsheimer RL, „ Struktura DNA bakteriofaga PhiX 174. III. Ultrawirówki potwierdzające strukturę pierścieniową. » , J. Mol. Biol. , vol. 5,1962, s. 424-434 ( PMID 13945085 )
- (w) Sanger, F., Air GM, BG Barrell, Brown NL, Coulson AR, CA Fiddes, Hutchison CA, Slocombe PM, Mr. Smith, „ Sekwencja nukleotydowa bakteriofaga phi X174 DNA. » , Nature , vol. 265,1977, s. 687-695 ( PMID 870828 )
- (w) Smith HO, Hutchison CA 3rd, Pfannkoch C. Venter JC, „ Generowanie syntetycznego genomu przez składanie całego genomu: bakteriofag {phi} X174 z syntetycznych oligonukleotydów. » , Proc. Natl. Acad. Sci. Stany Zjednoczone , vol. 100,2003, s. 15440-15445 ( PMID 14657399 )[1]
- {en} http://m.jbc.org/content/276/9/6093.full