Topienie o wysokiej rozdzielczości
Analiza krzywej topnienia wysokiej rozdzielczości lub wysokiej rozdzielczości stopu (HRM) jest techniką, w biologii molekularnej do wykrywania mutacji w polimorfizmu genetycznego i różnic epigenetyczne w próbkach DNA podwójnej nici. Został odkryty i opracowany przez Idaho Technology na Uniwersytecie Utah. Ma różne zalety w stosunku do innych technik genotypowania :
- ZZL kosztuje mniej niż inne techniki, takie jak sekwencjonowanie i Taqman . To sprawia, że jest to technika z wyboru w przypadku dużych projektów.
- Jest to stosunkowo szybkie i łatwe.
metoda
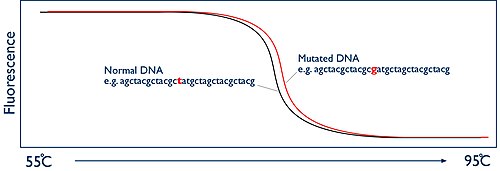
Analiza HRM jest wykonywana przy użyciu próbek dwuniciowego DNA. Region DNA potencjalnie niosący mutację jest najpierw amplifikowany przez PCR . Amplikon następnie ogrzewa się bardzo powoli, a dokładniej do około 50 do 95 ° C W pewnym momencie dwie nici DNA rozdzielają się - nazywa się to fuzją DNA. Zasada HRM polega na obserwowaniu tej fuzji w czasie rzeczywistym.
Jest to możliwe dzięki specjalnym związkom, zwanym barwnikami interkalującymi, które mają właściwość zwiększania fluorescencji po przyłączeniu do dwuniciowego DNA. W przypadku braku podwójnej nici nie mogą się przyczepiać, a ich poziom fluorescencji jest bardzo niski. Na początku analizy HRM, w najniższej temperaturze, intensywność fluorescencji jest bardzo wysoka, ponieważ cały amplikon występuje w postaci dwuniciowej. Jednak wraz ze wzrostem temperatury i rozdzielaniem się pasm fluorescencja spada. Rejestracja intensywności fluorescencji w funkcji temperatury nazywana jest krzywą topnienia.
Nawet niewielka zmiana w sekwencji DNA prowadzi do zmiany kinetyki fuzji, którą można wykryć dzięki zastosowanej wysokiej rozdzielczości.
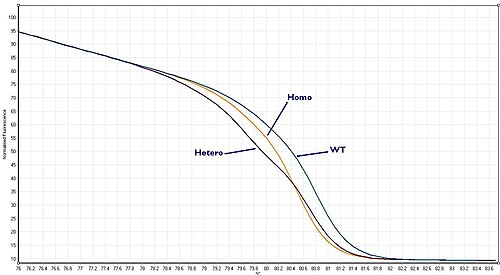
Typ dziki, heterozygotyczny lub homozygotyczny
Ponieważ podmiot ma dwie kopie każdego genu, istnieją trzy możliwości:
- Żaden allel nie zawiera mutacji (typ dziki)
- Oba allele zawierają mutację ( heterozygotyczna )
- Oba allele zawierają mutację ( homozygotyczne )
Podczas jednoczesnej amplifikacji dwóch alleli obserwowana krzywa fluorescencji odpowiada sumie krzywych fluorescencji dwóch alleli. Jeśli rozdzielczość jest wystarczająca, można je wyróżnić:
Aplikacje HRM
Genotypowanie SNP
SNP (polimorfizm pojedynczego nukleotydu ) to mutacje, które wpływają tylko na jeden nukleotyd. Można je wykryć za pomocą HRM, co jest znacznie tańsze niż użycie chipa DNA . Na przykład, podstawienie guaniny przez adeninę w genie VSSC ( napięcie kanał sodowy wrażliwy ) nadaje im odporność na permetryna , akarycyd w świerzb roztocza (odpowiedzialny za świerzb ). Ten SNP powoduje nieco niższą temperaturę topnienia DNA odpornych roztoczy. Dzieje się tak, ponieważ wiązanie GC jest silniejsze niż wiązanie A-T, ze względu na liczbę zaangażowanych wiązań wodorowych.
W diagnostyce klinicznej HRM jest czasami preferowane lub łączone z sekwencjonowaniem w celu identyfikacji mutacji (na przykład zidentyfikowano ponad 400 mutacji w genach BRCA1 i BRCA2, związanych z rakiem piersi). Obecnie HRM jest wykorzystywane głównie jako narzędzie przesiewowe i w razie potrzeby uzupełniane przez sekwencjonowanie.
Test zygotyczności
Test zygotyczności pozwala sprawdzić, czy dwoje ludzi to „prawdziwe” bliźniaki, czy bliźnięta po stronie braterskiej. Wśród wielu metod, które można zastosować, można wymienić HRM.
Epigenetyka
Metodologia HRM została również wykorzystana do analizy statusu metylacji DNA . Ma to szczególne znaczenie w onkogenetyce, ponieważ metylacja DNA może zmniejszyć ekspresję genów regulujących nowotwór, takich jak te, które regulują apoptozę i naprawę DNA. Ma również wpływ na odpowiedź na chemioterapię . Na przykład, jeśli promotor genu naprawy MGMT (metylotransferazy O-6-metyloguanino-DNA) jest metylowany, guz będzie mniej wrażliwy na czynniki alkilujące . W badaniu przeprowadzonym przez HRM na 19 przeanalizowanych raków jelita grubego 8 wykazało metylację promotora MGMT.
Aby uzyskać ten wynik, próbkę DNA traktuje się wodorosiarczynem , który ma właściwość przekształcania niemetylowanych cytozyn (i tylko tych) w uracyl . W ten sposób zmienia się sekwencja zasad, co wpływa na krzywą topnienia i może zostać wykryte podczas analizy HRM. HRM umożliwia poznanie proporcji metylacji w danej próbce, porównując krzywą topnienia z progresywnym zakresem utworzonym z mieszanin niemetylowanego DNA i całkowicie metylowanego DNA.
Zastosowano barwniki interkalujące
Pierwszym barwnikiem używanym w HRM jest SYBR Green . Fluoryzuje tylko po związaniu z dwuniciowym DNA. W wysokich stężeniach może hamować reakcję polimeryzacji podczas PCR. Jego użycie jest możliwe przy niskim stężeniu, ale nie jest zalecane przez niektórych badaczy, ze względu na zjawisko przeskakiwania barwnika : cząsteczki barwnika uwolnione w wyniku fuzji w pewnych punktach są ponownie wbudowywane w inne miejsce, gdzie nici nie są jeszcze rozdzielone, co prowadzi do błędu wyniki. Nowe barwniki nasycające, takie jak LC Green, LC Green Plus, ResoLight, EvaGreen, Chromofy i SYTO 9 są dostępne na rynku i wykazały swoją skuteczność w HRM. Jednak SYBR Green jest nadal używany z niektórymi urządzeniami.
Bibliografia
- Aby zapoznać się z historią HRM, patrz http://www.dna.utah.edu/Hi-Res/TOP_Hi-Res%20Melting.html
- Pasay C, Arlian L, Morgan M i wsp. , „ Analiza topnienia o wysokiej rozdzielczości w celu wykrycia mutacji związanej z opornością na permetrynę w populacji świerzbu ”, Med. Weterynarz. Entomol. , vol. 22, n o 1,Marzec 2008, s. 82–8 ( PMID 18380658 , DOI 10.1111 / j.1365-2915.2008.00716.x , czytaj online )
- James PA, Doherty R, Harris M i in. , „ Optymalna selekcja osobników do badania mutacji BRCA: porównanie dostępnych metod ”, J. Clin. Oncol. , vol. 24 N O 4,Luty 2006, s. 707-15 ( PMID 16446345 , DOI 10.1200 / JCO.2005.01.9737 , czytaj online )
- Krypuy M, Ahmed AA, Etemadmoghadam D, et al. , „ High resolution melting for mutation Scanning of TP53 exons 5-8 ”, BMC Cancer , vol. 7,2007, s. 168 ( PMID 17764544 , PMCID 2025602 , DOI 10.1186 / 1471-2407-7-168 , czytaj online )
- Wojdacz TK, Dobrovic A, „ Methylation-sensitive high resolution melting (MS-HRM): a new approach for sensitive and high-throughput assessment of methylation ”, Nucleic Acids Res. , vol. 35, n o 6,2007, e41 ( PMID 17289753 , PMCID 1874596 , DOI 10.1093 / nar / gkm013 , czytaj online )
- Reed GH, Kent JO, Wittwer CT, „ Analiza topnienia DNA o wysokiej rozdzielczości dla prostej i wydajnej diagnostyki molekularnej ”, Pharmacogenomics , tom. 8, N O 6,czerwiec 2007, s. 597–608 ( PMID 17559349 , DOI 10.2217 / 14622416.8.6.597 , czytaj online ) jako PDF
- Gundry CN, Vandersteen JG, Reed GH, Pryor RJ, Chen J, Wittwer CT, „ Amplicon melting analysis with marked primers : a closed-tube method for differentting homozygotes and heterozygotes ”, Clin. Chem. , vol. 49, n o 3,Marzec 2003, s. 396–406 ( PMID 12600951 , DOI 10.1373 / 49.3.396 , czytaj online )
- Pornprasert S, Phusua A, Suanta S, Saetung R, Sanguansermsri T, „ Detection of alpha-thalassemia-1 Southeast Asian type using real-time gap-PCR with SYBR Green1 and high resolution melting analysis ”, Eur. J. Haematol. , vol. 80, n o 6,czerwiec 2008, s. 510–4 ( PMID 18284625 , DOI 10.1111 / j.1600-0609.2008.01055.x , czytaj online )
- (fr) Ten artykuł jest częściowo lub w całości zaczerpnięty z artykułu w angielskiej Wikipedii zatytułowanego „ High Resolution Melt ” ( zobacz listę autorów ) .